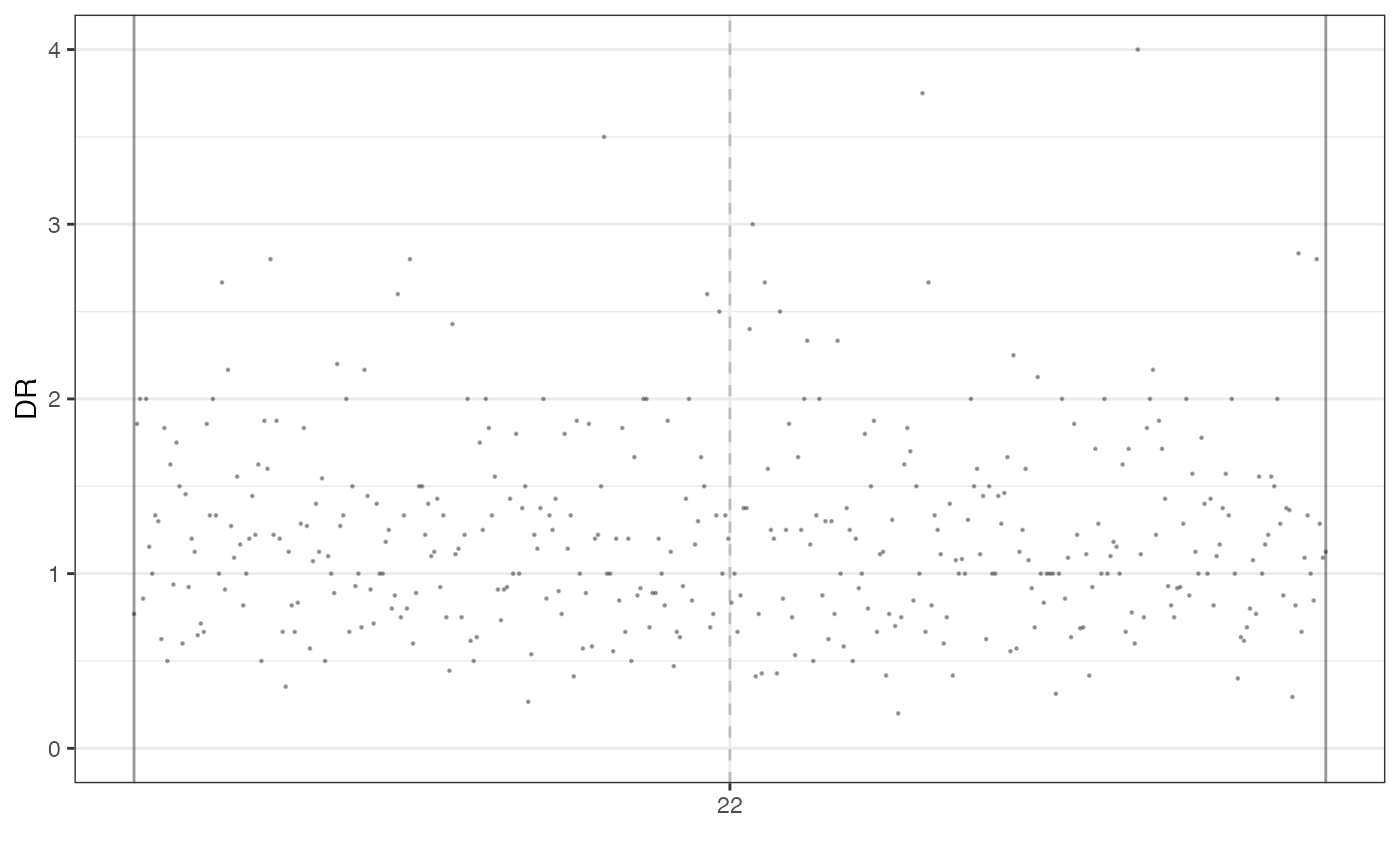
This function plots the genome-wide Depth Ratio (DR) of a specific sample.
Arguments
- seq_result
A data frame containing sequencing results.
- sample
The name of the sample for which the plot will be generated.
- chromosomes
A character vector specifying the chromosomes to include in the plot (default: NULL, i.e., all the chromosomes).
- N
The number of mutations to sample for plotting (default: 5000).
Examples
# set the seed of the random number generator
set.seed(0)
sim <- SpatialSimulation()
sim$add_mutant(name = "A",
growth_rates = 0.2,
death_rates = 0.0)
sim$place_cell("A", 500, 500)
sim$run_up_to_time(100)
#>
[████████████████████████████████████████] 100% [00m:00s] Saving snapshot
n_w <- n_h <- 10
ncells <- 0.8 * n_w * n_h
bbox <- sim$search_sample(c("A" = ncells), n_w, n_h)
sim$sample_cells("Sample", bbox$lower_corner, bbox$upper_corner)
forest <- sim$get_samples_forest()
m_engine <- MutationEngine(setup_code = "demo")
#> Downloading reference genome...
#> Reference genome downloaded
#> Decompressing reference file...done
#> Downloading SBS file...
#> SBS file downloaded
#> Downloading indel file...
#> indel file downloaded
#> Downloading driver mutation file...
#> Driver mutation file downloaded
#> Downloading passenger CNAs file...
#> Passenger CNAs file downloaded
#> Downloading germline mutations...
#> Germline mutations downloaded
#> Building context index...
#>
[█---------------------------------------] 0% [00m:00s] Processing chr. 22
[██████████------------------------------] 24% [00m:01s] Processing chr. 22
[████████████████████--------------------] 49% [00m:02s] Processing chr. 22
[██████████████████████████████----------] 73% [00m:03s] Processing chr. 22
[████████████████████████████████████████] 98% [00m:04s] Processing chr. 22
[████████████████████████████████████████] 100% [00m:04s] Context index built
#>
[█---------------------------------------] 0% [00m:00s] Saving context index
[████████████████████████████████████████] 100% [00m:00s] Context index saved
#> done
#> Building repeated sequence index...
#>
[█---------------------------------------] 0% [00m:00s] Processing chr. 22
[█---------------------------------------] 0% [00m:00s] Processing chr. 22
[█---------------------------------------] 0% [00m:01s] Processing chr. 22
[█---------------------------------------] 0% [00m:05s] Processing chr. 22
[█---------------------------------------] 0% [00m:07s] Processing chr. 22
[█---------------------------------------] 0% [00m:09s] Processing chr. 22
[█---------------------------------------] 0% [00m:10s] Processing chr. 22
[█---------------------------------------] 0% [00m:12s] Processing chr. 22
[█---------------------------------------] 0% [00m:18s] Processing chr. 22
[████████████████████████████████████████] 100% [00m:18s] RS index built
#>
[█---------------------------------------] 0% [00m:00s] Saving RS index
[█---------------------------------------] 0% [00m:00s] Saving RS index
[█---------------------------------------] 0% [00m:01s] Saving RS index
[████████████████------------------------] 39% [00m:03s] Saving RS index
[███████████████████████████████---------] 76% [00m:04s] Saving RS index
done
#>
[████████████████████████████████████████] 100% [00m:04s] RS index saved
#>
[█---------------------------------------] 0% [00m:00s] Loading germline
[████████████████████████████████████████] 100% [00m:01s] Loading germline
#>
[████████████████████████████████████████] 100% [00m:01s] Germline loaded
#>
[█---------------------------------------] 0% [00m:00s] Saving germline
[████████████████████████████████████████] 100% [00m:00s] Germline saved
m_engine$add_mutant(mutant_name="A", passenger_rates=c(SNV=5e-8))
#>
[█---------------------------------------] 0% [00m:00s] Retrieving "A" SNVs
[████████████████████████████████████████] 100% [00m:00s] "A" SNVs retrieved
m_engine$add_exposure(c(SBS1 = 0.2, SBS5 = 0.8))
phylo_forest <- m_engine$place_mutations(forest, 10, 10)
#>
[█---------------------------------------] 0% [00m:00s] Placing mutations
[████████████████████████████████████████] 100% [00m:00s] Mutations placed
seq_results <- simulate_seq(
phylo_forest,
chromosomes = c('22'),
coverage = 10,
write_SAM = F
)
#>
[█---------------------------------------] 0% [00m:00s] Found 22
[█---------------------------------------] 0% [00m:00s] Reading 22
[█---------------------------------------] 0% [00m:01s] Reading 22
[█---------------------------------------] 0% [00m:02s] Reading 22
[█---------------------------------------] 0% [00m:03s] Reading 22
[█---------------------------------------] 0% [00m:03s] Processing chr. 22
[█---------------------------------------] 1% [00m:04s] Processing chr. 22
[██--------------------------------------] 3% [00m:05s] Processing chr. 22
[███-------------------------------------] 5% [00m:06s] Processing chr. 22
[███-------------------------------------] 6% [00m:07s] Processing chr. 22
[████------------------------------------] 8% [00m:08s] Processing chr. 22
[█████-----------------------------------] 10% [00m:09s] Processing chr. 22
[█████-----------------------------------] 12% [00m:10s] Processing chr. 22
[██████----------------------------------] 14% [00m:11s] Processing chr. 22
[███████---------------------------------] 16% [00m:12s] Processing chr. 22
[████████--------------------------------] 19% [00m:13s] Processing chr. 22
[█████████-------------------------------] 21% [00m:14s] Processing chr. 22
[██████████------------------------------] 23% [00m:15s] Processing chr. 22
[███████████-----------------------------] 25% [00m:16s] Processing chr. 22
[███████████-----------------------------] 27% [00m:17s] Processing chr. 22
[████████████----------------------------] 29% [00m:18s] Processing chr. 22
[█████████████---------------------------] 31% [00m:19s] Processing chr. 22
[██████████████--------------------------] 33% [00m:20s] Processing chr. 22
[██████████████--------------------------] 34% [00m:21s] Processing chr. 22
[███████████████-------------------------] 37% [00m:22s] Processing chr. 22
[████████████████------------------------] 39% [00m:23s] Processing chr. 22
[█████████████████-----------------------] 41% [00m:24s] Processing chr. 22
[██████████████████----------------------] 43% [00m:25s] Processing chr. 22
[███████████████████---------------------] 45% [00m:26s] Processing chr. 22
[███████████████████---------------------] 46% [00m:27s] Processing chr. 22
[████████████████████--------------------] 48% [00m:28s] Processing chr. 22
[█████████████████████-------------------] 50% [00m:29s] Processing chr. 22
[█████████████████████-------------------] 52% [00m:30s] Processing chr. 22
[██████████████████████------------------] 54% [00m:31s] Processing chr. 22
[███████████████████████-----------------] 56% [00m:32s] Processing chr. 22
[████████████████████████----------------] 58% [00m:33s] Processing chr. 22
[█████████████████████████---------------] 60% [00m:34s] Processing chr. 22
[█████████████████████████---------------] 62% [00m:35s] Processing chr. 22
[███████████████████████████-------------] 65% [00m:36s] Processing chr. 22
[███████████████████████████-------------] 67% [00m:37s] Processing chr. 22
[████████████████████████████------------] 69% [00m:38s] Processing chr. 22
[█████████████████████████████-----------] 70% [00m:39s] Processing chr. 22
[█████████████████████████████-----------] 71% [00m:40s] Processing chr. 22
[██████████████████████████████----------] 73% [00m:41s] Processing chr. 22
[███████████████████████████████---------] 75% [00m:42s] Processing chr. 22
[███████████████████████████████---------] 77% [00m:43s] Processing chr. 22
[████████████████████████████████--------] 79% [00m:44s] Processing chr. 22
[█████████████████████████████████-------] 81% [00m:45s] Processing chr. 22
[██████████████████████████████████------] 83% [00m:46s] Processing chr. 22
[███████████████████████████████████-----] 85% [00m:47s] Processing chr. 22
[████████████████████████████████████----] 88% [00m:48s] Processing chr. 22
[█████████████████████████████████████---] 90% [00m:49s] Processing chr. 22
[█████████████████████████████████████---] 92% [00m:50s] Processing chr. 22
[██████████████████████████████████████--] 94% [00m:51s] Processing chr. 22
[███████████████████████████████████████-] 96% [00m:52s] Processing chr. 22
[████████████████████████████████████████] 98% [00m:53s] Processing chr. 22
[████████████████████████████████████████] 98% [00m:54s] Processing chr. 22
[████████████████████████████████████████] 99% [00m:55s] Processing chr. 22
[████████████████████████████████████████] 99% [00m:56s] Processing chr. 22
[████████████████████████████████████████] 99% [00m:57s] Processing chr. 22
[████████████████████████████████████████] 99% [00m:58s] Processing chr. 22
[████████████████████████████████████████] 99% [00m:59s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:00s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:01s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:02s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:03s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:04s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:05s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:06s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:07s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:08s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:09s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:10s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:11s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:12s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:13s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:14s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:15s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:16s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:17s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:18s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:19s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:20s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:21s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:22s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:23s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:24s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:25s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:26s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:27s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:28s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:29s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:30s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:31s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:32s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:33s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:34s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:35s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:36s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:37s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:38s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:39s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:40s] Processing chr. 22
[████████████████████████████████████████] 99% [01m:41s] Processing chr. 22
[████████████████████████████████████████] 100% [01m:43s] Reads simulated
library(dplyr)
# filter germinal mutations
f_seq <- seq_results$mutations %>% filter(classes!="germinal")
plot_DR(f_seq, sample="Sample")
 unlink('demo', recursive = T)
unlink('demo', recursive = T)