Disclaimer: RACES/ProCESS internally implements the probability distributions using the C++11 random number distribution classes. The standard does not specify their algorithms, and the class implementations are left free for the compiler. Thus, the simulation output depends on the compiler used to compile RACES, and because of that, the results reported in this article may differ from those obtained by the reader.
We build a tumour with a single mutant “A” and grow it up to 10 cells.
library(ProCESS)
# set the seed of the random number generator for repeatability
set.seed(0)
sim <- TissueSimulation(width = 600, height = 600)
# set the delta time between two time series samples
sim$history_delta <- 10
# avoid drift
sim$death_activation_level <- 50
sim$add_mutant(name = "A", growth_rates = 0.12, death_rates = 0.05)
sim$place_cell("A", 300, 300)
sim$run_up_to_size("A", 10)
#> [████████████████████████████████████████] 100% [00m:00s] Saving snapshotWe add a new mutant “B” as sub-clone of “A” and let the simulation evolve until “B” reaches 30 cells.
sim$add_mutant(name = "B", growth_rates = 0.145, death_rates = 0.06)
sim$mutate_progeny(sim$choose_cell_in("A"), "B")
sim$run_up_to_size("B", 30)
#> [████████████████████████████████████████] 100% [00m:00s] Saving snapshotWe add a further mutants “C” as sub-clones for “A” and let the tumour grow again up to the point in which “C” consists of 25000 cells.
sim$add_mutant(name = "C", growth_rates = 0.15, death_rates = 0.06)
sim$mutate_progeny(sim$choose_cell_in("A"), "C")
sim$run_up_to_size("C", 25000)
#> [█████████-------------------------------] 21% [00m:00s] Cells: 18972 [███████████████-------------------------] 36% [00m:00s] Cells: 30162 [████████████████████--------------------] 49% [00m:01s] Cells: 39221 [█████████████████████████---------------] 60% [00m:02s] Cells: 46563 [█████████████████████████████-----------] 71% [00m:04s] Cells: 53905 [█████████████████████████████████-------] 80% [00m:05s] Cells: 60371 [████████████████████████████████████----] 89% [00m:06s] Cells: 65934 [███████████████████████████████████████-] 97% [00m:07s] Cells: 71314 [████████████████████████████████████████] 100% [00m:07s] Saving snapshotThen, we plot the tissue and the simulation state.
plot_tissue(sim)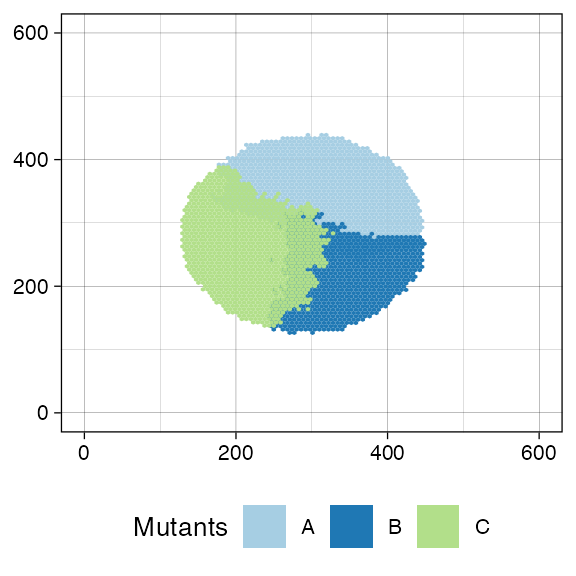
plot_state(sim)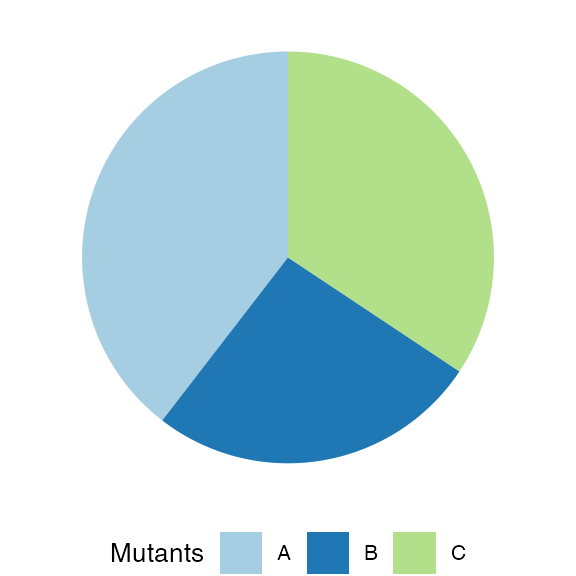
Tissue sampling level
We can collect three samples: “S1” and “S2”.
We can plot the tissue simulation after the sampling. We label the sampled region to improve readability, but it is not mandatory.
plot_tissue(sim) +
ggplot2::annotate("text", x = (145 + 215) / 2, y = (230 + 300) / 2,
label = "S1", parse = TRUE) +
ggplot2::annotate("text", x = (350 + 420) / 2, y = (300 + 370) / 2,
label = "S2", parse = TRUE)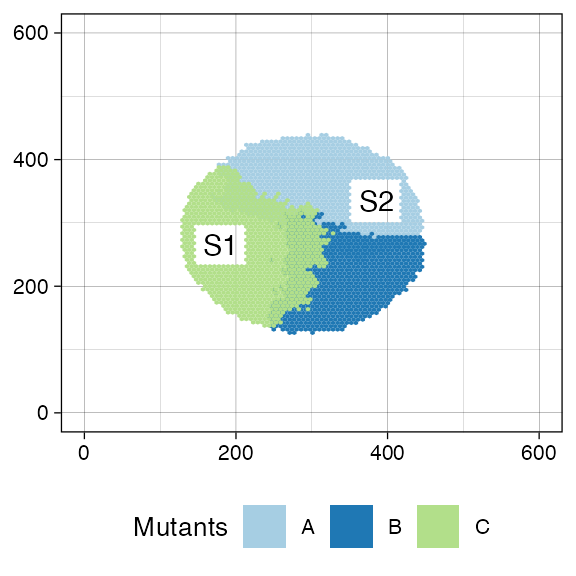
After the sampling, we can add a new mutant “D” as sub-clone of “B”, let the simulation further evolve until the sum of the cardinalities of the mutants “C” and “D” is 100000, and sample the tissue again.
sim$add_mutant(name = "D", growth_rates = 0.8, death_rates = 0.01)
sim$mutate_progeny(sim$choose_cell_in("B", c(240, 130), c(250, 150)), "D")
sim$run_until(sim$var("C") + sim$var("D") == 100000)
#> [██--------------------------------------] 4% [00m:00s] Cells: 70006 [████------------------------------------] 8% [00m:00s] Cells: 77450 [██████----------------------------------] 13% [00m:01s] Cells: 84657 [████████--------------------------------] 19% [00m:02s] Cells: 92078 [███████████-----------------------------] 26% [00m:03s] Cells: 99533 [██████████████--------------------------] 33% [00m:04s] Cells: 107442 [█████████████████-----------------------] 40% [00m:05s] Cells: 114957 [████████████████████--------------------] 48% [00m:06s] Cells: 122828 [███████████████████████-----------------] 56% [00m:07s] Cells: 130598 [██████████████████████████--------------] 64% [00m:08s] Cells: 137537 [█████████████████████████████-----------] 70% [00m:09s] Cells: 143776 [████████████████████████████████--------] 78% [00m:10s] Cells: 150298 [███████████████████████████████████-----] 85% [00m:11s] Cells: 157062 [█████████████████████████████████████---] 92% [00m:12s] Cells: 163108 [████████████████████████████████████████] 98% [00m:13s] Cells: 168497 [████████████████████████████████████████] 100% [00m:14s] Saving snapshot
sim$sample_cells("S3", c(360, 80), c(430, 150))
plot_tissue(sim) +
ggplot2::annotate("text", x = (360 + 430) / 2, y = (80 + 150) / 2,
label = "S3", parse = TRUE)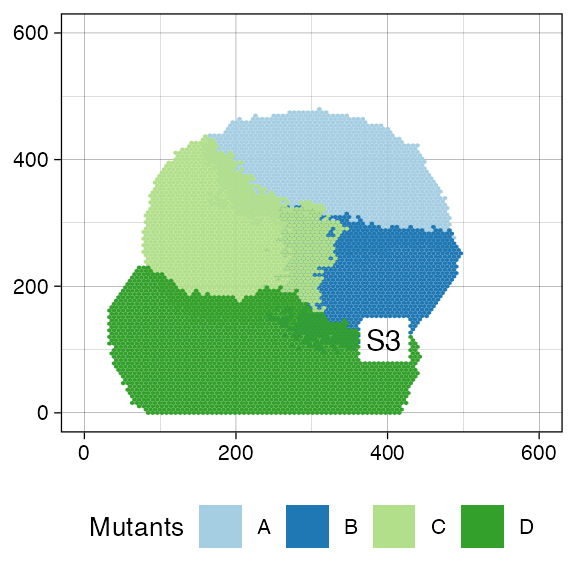
plot_state(sim)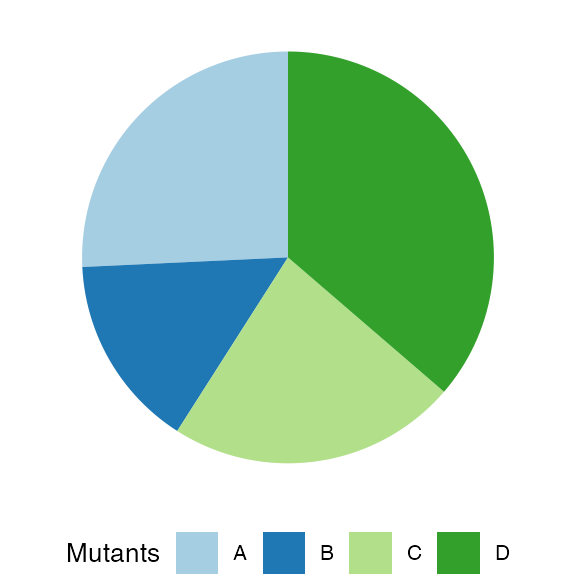
The time-series plot represents the species cardinalities along the simulation.
plot_timeseries(sim)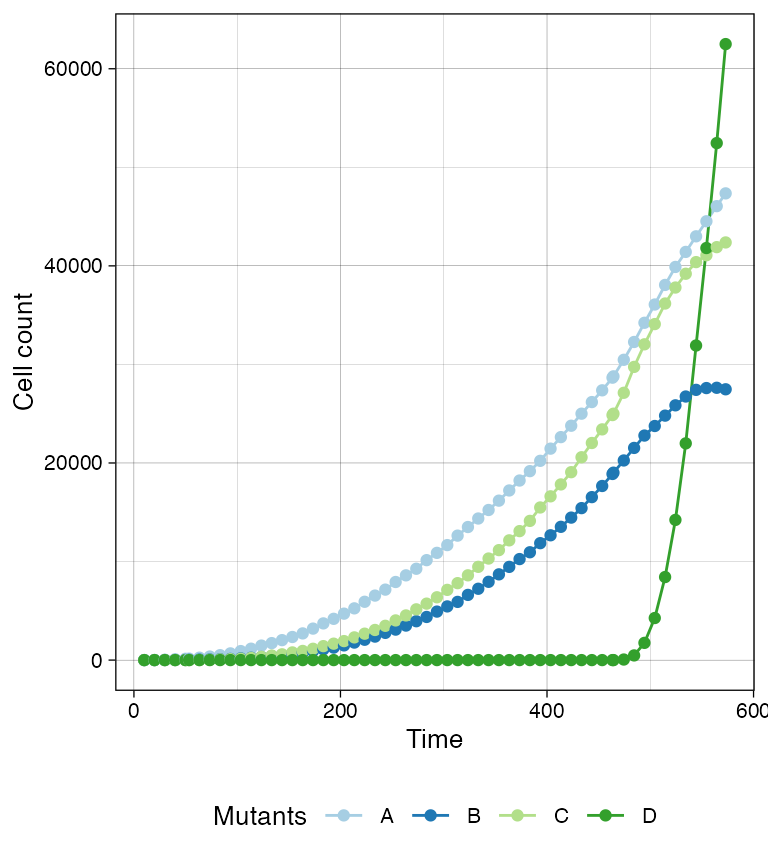
The Muller plot, instead, overviews the percentage of cells in each species along the simulation evolution.
plot_muller(sim)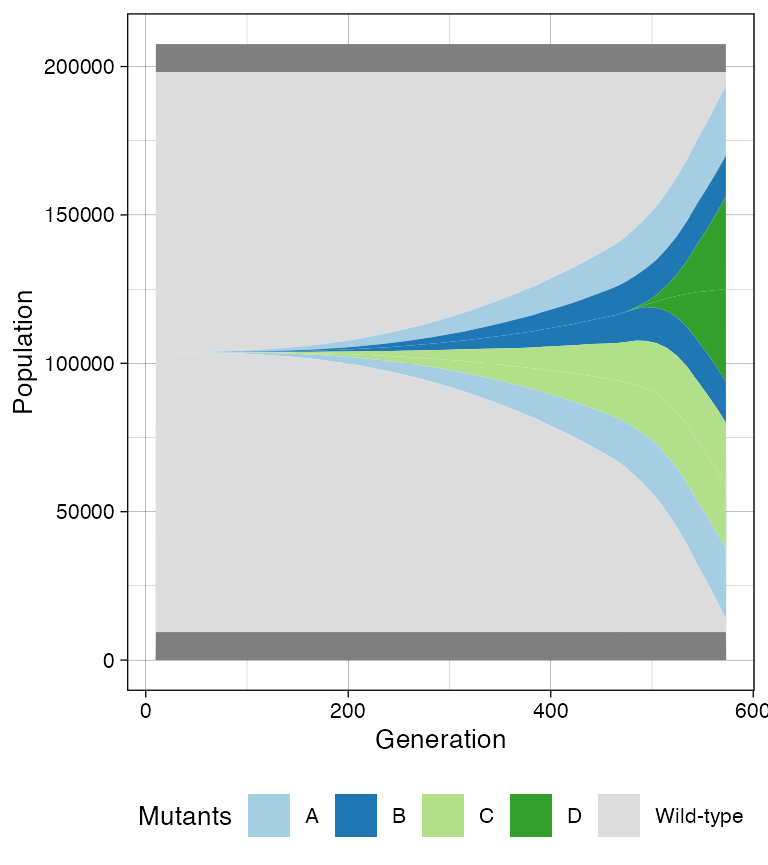
We can build the sample forest and plot it.
sample_forest <- sim$get_sample_forest()
library(dplyr)
#>
#> Attaching package: 'dplyr'
#> The following objects are masked from 'package:stats':
#>
#> filter, lag
#> The following objects are masked from 'package:base':
#>
#> intersect, setdiff, setequal, union
plot_forest(sample_forest) %>%
annotate_forest(sample_forest)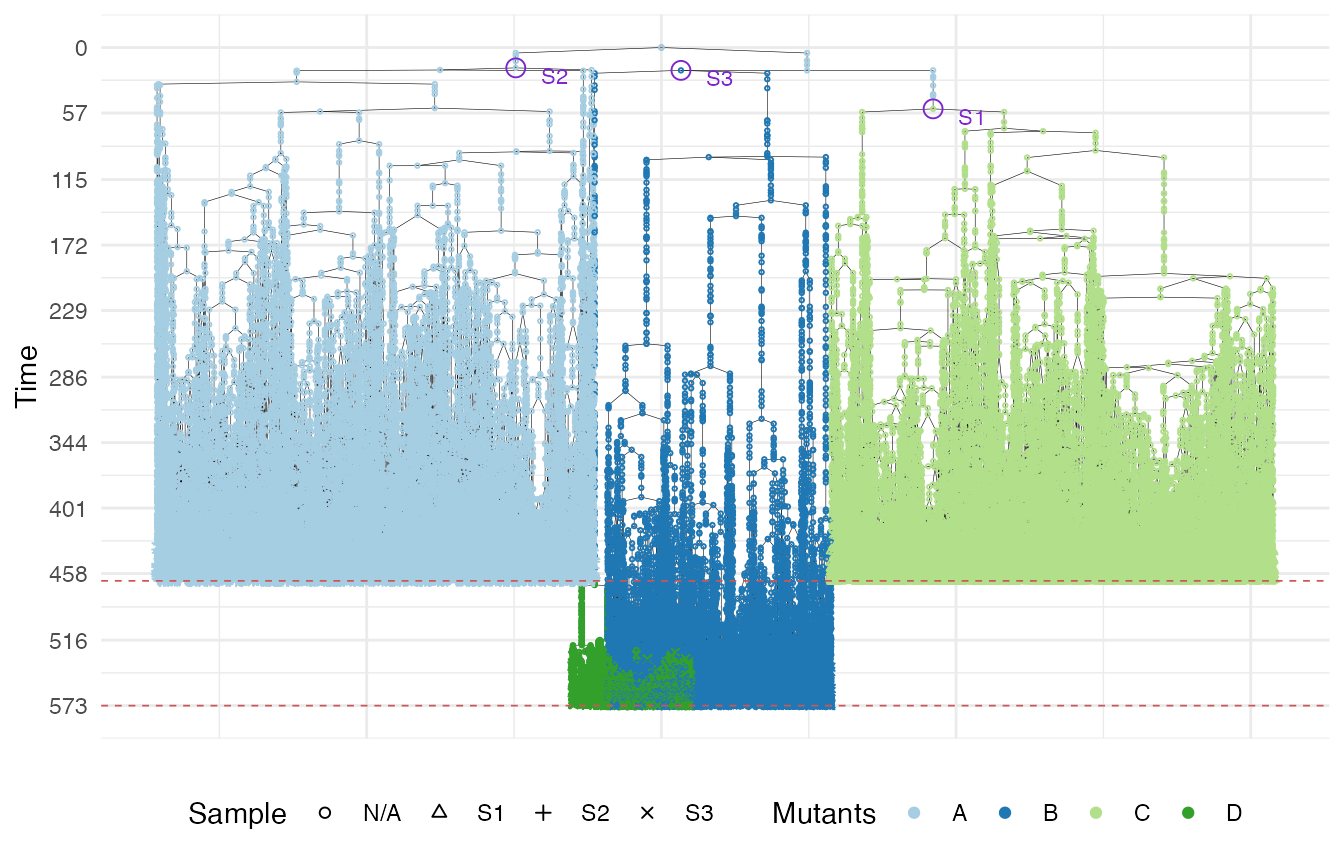
Cell genetic characterisation level
We first build the mutation engine to label each cell in the sample forest. The mutation engine pre-defined setups automatically download all the needed file. All the pre-configured setups, but “demo” requires a COSMIC account to download the signature files from the COSMIC site.
# build the mutation engine according to the pre-defined setup "demo"
m_engine <- MutationEngine(setup_code="demo",
COSMIC_account = list(email = "foo@bar.com",
password = "#########"))
#> [█---------------------------------------] 0% [00m:00s] Loading context index [████████████████████████████████████████] 100% [00m:00s] Context index loaded
#> [█---------------------------------------] 0% [00m:00s] Loading RS index [█████████████████-----------------------] 42% [00m:01s] Loading RS index [█████████████████████████████████-------] 81% [00m:02s] Loading RS index [████████████████████████████████████████] 100% [00m:02s] RS index loaded
#> [█---------------------------------------] 0% [00m:00s] Loading germline [████████████████████████████████████████] 100% [00m:00s] Germline loadedWe must genomically characterise the tissue simulation mutants providing passenger mutation rates and the list of driver mutations.
# genetically characterise the mutants
m_engine$add_mutant("A", passenger_rates = c(SNV = 2e-9, indel = 2e-9),
drivers = list(Mutation("22", 16085675, "GCCTCCCGA", "G"),
CNA("D", "22", 22453799, 200000,
allele = 1)))
#> [█---------------------------------------] 0% [00m:00s] Retrieving "A" SIDs [█---------------------------------------] 0% [00m:00s] Found 22 [█---------------------------------------] 0% [00m:00s] Reading 22 [████████--------------------------------] 18% [00m:01s] Reading 22 [███████████████-------------------------] 36% [00m:02s] Reading 22 [██████████████████████------------------] 52% [00m:03s] Reading 22 [█████████████████████████████-----------] 71% [00m:04s] Reading 22 [████████████████████████████████████----] 89% [00m:05s] Reading 22 [████████████████████████████████████████] 100% [00m:05s] "A"'s SIDs validated
m_engine$add_mutant("B", passenger_rates = c(SNV = 1e-9, indel = 2e-9,
CNA = 1e-9),
drivers = list(SNV("22", 16050185, "C", allele = 1),
CNA(type = "A", chr = "22",
chr_pos = 16485130, len = 200000)))
#> [█---------------------------------------] 0% [00m:00s] Retrieving "B" SIDs [████████████████████████████████████████] 100% [00m:00s] "B"'s SIDs validated
m_engine$add_mutant("C", passenger_rates = list(SNV = 2e-8, indel = 2e-9),
drivers = list(SNV("22", 32786322, "G"),
list("DGCR8 P26L", allele = 1)))
#> [█---------------------------------------] 0% [00m:00s] Retrieving "C" SIDs [████████████████████████████████████████] 100% [00m:00s] "C"'s SIDs validated
m_engine$add_mutant("D", passenger_rates = list(SNV = 2e-9, indel = 2e-9),
drivers = list(SNV("22", 51240420, "T")))
#> [█---------------------------------------] 0% [00m:00s] Retrieving "D" SIDs [████████████████████████████████████████] 100% [00m:00s] "D"'s SIDs validatedWe also need to declare the exposures along the simulation.
# add SNV and indel default exposures. This will be used from simulated time 0
# up to the successive exposure change.
m_engine$add_exposure(coefficients = c(SBS13 = 0.2, SBS1 = 0.8))
m_engine$add_exposure(c(ID2 = 0.6, ID13 = 0.2, ID21 = 0.2))
# add a new SNV exposure that will be used from simulated
# time 100 up to the successive exposure change.
m_engine$add_exposure(time = 100, c(SBS5 = 0.3, SBS2 = 0.2, SBS3 = 0.5))
m_engine$add_exposure(time = 120, c(SBS5 = 0.3, SBS2 = 0.2, SBS3 = 0.5,
ID1 = 0.8, ID9 = 0.2))
m_engine
#> MutationEngine
#> Passenger rates
#> "A": {SNV: 2e-09, indel: 2e-09}
#> "B": {SNV: 1e-09, indel: 2e-09, CNA: 1e-09}
#> "C": {SNV: 2e-08, indel: 2e-09}
#> "D": {SNV: 2e-09, indel: 2e-09}
#>
#> Driver mutations
#> "A":
#> (chr22(16085675)[GCCTCCCGA>G]) on random allele
#> CNA("D",chr22(22453799), len: 200000, allele: 1)
#> "B":
#> (chr22(16050185)[A>C]) on allele 1
#> CNA("A",chr22(16485130), len: 200000)
#> "C":
#> (chr22(32786322)[T>G]) on random allele
#> DGCR8 P26L (chr22(20073563)[C>T]) on allele 1
#> "D":
#> (chr22(51240420)[G>T]) on random allele
#>
#> Timed Exposure
#> SBS Timed Exposures
#> [0, 100[: {"SBS1": 0.8, "SBS13": 0.2}
#> [100, 120[: {"SBS2": 0.2, "SBS3": 0.5, "SBS5": 0.3}
#> [120, ∞[: {"SBS2": 0.2, "SBS3": 0.5, "SBS5": 0.3}
#>
#> indel Timed Exposures
#> [0, 120[: {"ID13": 0.2, "ID2": 0.6, "ID21": 0.2}
#> [120, ∞[: {"ID1": 0.8, "ID9": 0.2}We are now ready to build the phylogenetic forest.
# place mutations on the sample forest assuming 1000 pre-neoplastic SNVs and
# 500 indels
phylo_forest <- m_engine$place_mutations(sample_forest, 1000, 500)
#> [█---------------------------------------] 0% [00m:00s] Placing mutations [█████████████████-----------------------] 42% [00m:01s] Placing mutations [█████████████████████████████-----------] 71% [00m:02s] Placing mutations [█████████████████████████████████-------] 81% [00m:03s] Placing mutations [█████████████████████████████████████---] 91% [00m:04s] Placing mutations [████████████████████████████████████████] 100% [00m:04s] Mutations placedThe phylogenetic forest maintains the genome mutations (SBSs, indels, and CNAs) of all the sampled cells.
mutations <- phylo_forest$get_sampled_cell_mutations()
head(mutations)
#> cell_id chr chr_pos allele ref alt type cause class
#> 1 683005 22 16080424 0 GTA G indel ID1 pre-neoplastic
#> 2 683005 22 16129596 0 T A SNV SBS1 pre-neoplastic
#> 3 683005 22 16130490 0 G A SNV SBS1 pre-neoplastic
#> 4 683005 22 16131174 0 A G SNV SBS1 pre-neoplastic
#> 5 683005 22 16156621 0 T TG indel ID1 pre-neoplastic
#> 6 683005 22 16192291 0 TC T indel ID1 pre-neoplastic
CNAs <- phylo_forest$get_sampled_cell_CNAs()
head(CNAs)
#> cell_id type chr begin end allele src.allele class
#> 1 683005 D 22 22453799 22653798 1 NA driver
#> 2 695907 D 22 22453799 22653798 1 NA driver
#> 3 695922 D 22 22453799 22653798 1 NA driver
#> 4 717557 D 22 22453799 22653798 1 NA driver
#> 5 724240 D 22 22453799 22653798 1 NA driver
#> 6 724361 D 22 22453799 22653798 1 NA driverSequencing level
We can simulate the sequencing of the collected samples by using the phylogenetic forest. For each SBS and indel mutation in the phylogenetic forest, the output reports the number of occurrences in the simulated reads, the coverage of each mutation locus, and the corresponding VAF in each of the samples.
seq_results <- simulate_seq(phylo_forest, coverage = 30,
with_normal_sample = FALSE)
#> [█---------------------------------------] 0% [00m:00s] [█---------------------------------------] 0% [00m:00s] Processing chr. 22 [█---------------------------------------] 0% [00m:04s] Processing chr. 22 [█---------------------------------------] 0% [00m:05s] Processing chr. 22 [█---------------------------------------] 0% [00m:06s] Processing chr. 22 [█---------------------------------------] 1% [00m:07s] Processing chr. 22 [█---------------------------------------] 1% [00m:08s] Processing chr. 22 [█---------------------------------------] 1% [00m:09s] Processing chr. 22 [█---------------------------------------] 2% [00m:10s] Processing chr. 22 [█---------------------------------------] 2% [00m:11s] Processing chr. 22 [██--------------------------------------] 3% [00m:12s] Processing chr. 22 [██--------------------------------------] 3% [00m:13s] Processing chr. 22 [██--------------------------------------] 3% [00m:14s] Processing chr. 22 [██--------------------------------------] 4% [00m:15s] Processing chr. 22 [██--------------------------------------] 4% [00m:16s] Processing chr. 22 [██--------------------------------------] 4% [00m:17s] Processing chr. 22 [███-------------------------------------] 5% [00m:18s] Processing chr. 22 [███-------------------------------------] 5% [00m:19s] Processing chr. 22 [███-------------------------------------] 6% [00m:20s] Processing chr. 22 [███-------------------------------------] 6% [00m:21s] Processing chr. 22 [███-------------------------------------] 6% [00m:22s] Processing chr. 22 [███-------------------------------------] 7% [00m:23s] Processing chr. 22 [███-------------------------------------] 7% [00m:24s] Processing chr. 22 [████------------------------------------] 8% [00m:25s] Processing chr. 22 [████------------------------------------] 8% [00m:26s] Processing chr. 22 [████------------------------------------] 8% [00m:27s] Processing chr. 22 [████------------------------------------] 9% [00m:28s] Processing chr. 22 [████------------------------------------] 9% [00m:29s] Processing chr. 22 [█████-----------------------------------] 10% [00m:30s] Processing chr. 22 [█████-----------------------------------] 10% [00m:31s] Processing chr. 22 [█████-----------------------------------] 10% [00m:32s] Processing chr. 22 [█████-----------------------------------] 11% [00m:33s] Processing chr. 22 [█████-----------------------------------] 11% [00m:34s] Processing chr. 22 [█████-----------------------------------] 11% [00m:35s] Processing chr. 22 [█████-----------------------------------] 12% [00m:36s] Processing chr. 22 [█████-----------------------------------] 12% [00m:37s] Processing chr. 22 [██████----------------------------------] 13% [00m:38s] Processing chr. 22 [██████----------------------------------] 13% [00m:39s] Processing chr. 22 [██████----------------------------------] 13% [00m:40s] Processing chr. 22 [██████----------------------------------] 14% [00m:41s] Processing chr. 22 [██████----------------------------------] 14% [00m:42s] Processing chr. 22 [███████---------------------------------] 15% [00m:43s] Processing chr. 22 [███████---------------------------------] 15% [00m:44s] Processing chr. 22 [███████---------------------------------] 15% [00m:45s] Processing chr. 22 [███████---------------------------------] 16% [00m:46s] Processing chr. 22 [███████---------------------------------] 16% [00m:47s] Processing chr. 22 [███████---------------------------------] 16% [00m:48s] Processing chr. 22 [███████---------------------------------] 17% [00m:49s] Processing chr. 22 [███████---------------------------------] 17% [00m:50s] Processing chr. 22 [████████--------------------------------] 18% [00m:51s] Processing chr. 22 [████████--------------------------------] 18% [00m:52s] Processing chr. 22 [████████--------------------------------] 18% [00m:53s] Processing chr. 22 [████████--------------------------------] 19% [00m:54s] Processing chr. 22 [████████--------------------------------] 19% [00m:55s] Processing chr. 22 [█████████-------------------------------] 20% [00m:56s] Processing chr. 22 [█████████-------------------------------] 20% [00m:57s] Processing chr. 22 [█████████-------------------------------] 20% [00m:58s] Processing chr. 22 [█████████-------------------------------] 21% [00m:59s] Processing chr. 22 [█████████-------------------------------] 21% [01m:00s] Processing chr. 22 [█████████-------------------------------] 22% [01m:01s] Processing chr. 22 [█████████-------------------------------] 22% [01m:02s] Processing chr. 22 [█████████-------------------------------] 22% [01m:03s] Processing chr. 22 [██████████------------------------------] 23% [01m:04s] Processing chr. 22 [██████████------------------------------] 23% [01m:05s] Processing chr. 22 [██████████------------------------------] 23% [01m:06s] Processing chr. 22 [██████████------------------------------] 24% [01m:07s] Processing chr. 22 [██████████------------------------------] 24% [01m:08s] Processing chr. 22 [███████████-----------------------------] 25% [01m:09s] Processing chr. 22 [███████████-----------------------------] 25% [01m:10s] Processing chr. 22 [███████████-----------------------------] 25% [01m:11s] Processing chr. 22 [███████████-----------------------------] 26% [01m:12s] Processing chr. 22 [███████████-----------------------------] 26% [01m:13s] Processing chr. 22 [███████████-----------------------------] 26% [01m:14s] Processing chr. 22 [███████████-----------------------------] 27% [01m:15s] Processing chr. 22 [███████████-----------------------------] 27% [01m:16s] Processing chr. 22 [████████████----------------------------] 28% [01m:17s] Processing chr. 22 [████████████----------------------------] 28% [01m:18s] Processing chr. 22 [████████████----------------------------] 28% [01m:19s] Processing chr. 22 [████████████----------------------------] 29% [01m:20s] Processing chr. 22 [████████████----------------------------] 29% [01m:21s] Processing chr. 22 [█████████████---------------------------] 30% [01m:22s] Processing chr. 22 [█████████████---------------------------] 30% [01m:23s] Processing chr. 22 [█████████████---------------------------] 30% [01m:24s] Processing chr. 22 [█████████████---------------------------] 31% [01m:25s] Processing chr. 22 [█████████████---------------------------] 31% [01m:26s] Processing chr. 22 [█████████████---------------------------] 31% [01m:27s] Processing chr. 22 [█████████████---------------------------] 32% [01m:28s] Processing chr. 22 [█████████████---------------------------] 32% [01m:29s] Processing chr. 22 [██████████████--------------------------] 33% [01m:30s] Processing chr. 22 [██████████████--------------------------] 33% [01m:31s] Processing chr. 22 [██████████████--------------------------] 33% [01m:32s] Processing chr. 22 [██████████████--------------------------] 34% [01m:33s] Processing chr. 22 [██████████████--------------------------] 34% [01m:34s] Processing chr. 22 [███████████████-------------------------] 35% [01m:35s] Processing chr. 22 [███████████████-------------------------] 35% [01m:36s] Processing chr. 22 [███████████████-------------------------] 35% [01m:37s] Processing chr. 22 [███████████████-------------------------] 36% [01m:38s] Processing chr. 22 [███████████████-------------------------] 36% [01m:39s] Processing chr. 22 [███████████████-------------------------] 36% [01m:40s] Processing chr. 22 [███████████████-------------------------] 37% [01m:41s] Processing chr. 22 [███████████████-------------------------] 37% [01m:42s] Processing chr. 22 [████████████████------------------------] 38% [01m:43s] Processing chr. 22 [████████████████------------------------] 38% [01m:44s] Processing chr. 22 [████████████████------------------------] 38% [01m:45s] Processing chr. 22 [████████████████------------------------] 39% [01m:46s] Processing chr. 22 [████████████████------------------------] 39% [01m:47s] Processing chr. 22 [█████████████████-----------------------] 40% [01m:48s] Processing chr. 22 [█████████████████-----------------------] 40% [01m:49s] Processing chr. 22 [█████████████████-----------------------] 40% [01m:50s] Processing chr. 22 [█████████████████-----------------------] 41% [01m:51s] Processing chr. 22 [█████████████████-----------------------] 41% [01m:52s] Processing chr. 22 [█████████████████-----------------------] 41% [01m:53s] Processing chr. 22 [█████████████████-----------------------] 42% [01m:54s] Processing chr. 22 [█████████████████-----------------------] 42% [01m:55s] Processing chr. 22 [██████████████████----------------------] 43% [01m:56s] Processing chr. 22 [██████████████████----------------------] 43% [01m:57s] Processing chr. 22 [██████████████████----------------------] 43% [01m:58s] Processing chr. 22 [██████████████████----------------------] 44% [01m:59s] Processing chr. 22 [██████████████████----------------------] 44% [02m:00s] Processing chr. 22 [██████████████████----------------------] 44% [02m:01s] Processing chr. 22 [███████████████████---------------------] 45% [02m:02s] Processing chr. 22 [███████████████████---------------------] 45% [02m:03s] Processing chr. 22 [███████████████████---------------------] 46% [02m:04s] Processing chr. 22 [███████████████████---------------------] 46% [02m:05s] Processing chr. 22 [███████████████████---------------------] 46% [02m:06s] Processing chr. 22 [███████████████████---------------------] 47% [02m:07s] Processing chr. 22 [███████████████████---------------------] 47% [02m:08s] Processing chr. 22 [███████████████████---------------------] 47% [02m:09s] Processing chr. 22 [████████████████████--------------------] 48% [02m:10s] Processing chr. 22 [████████████████████--------------------] 48% [02m:11s] Processing chr. 22 [████████████████████--------------------] 49% [02m:12s] Processing chr. 22 [████████████████████--------------------] 49% [02m:13s] Processing chr. 22 [████████████████████--------------------] 49% [02m:14s] Processing chr. 22 [█████████████████████-------------------] 50% [02m:15s] Processing chr. 22 [█████████████████████-------------------] 50% [02m:16s] Processing chr. 22 [█████████████████████-------------------] 51% [02m:17s] Processing chr. 22 [█████████████████████-------------------] 51% [02m:18s] Processing chr. 22 [█████████████████████-------------------] 51% [02m:19s] Processing chr. 22 [█████████████████████-------------------] 52% [02m:20s] Processing chr. 22 [█████████████████████-------------------] 52% [02m:21s] Processing chr. 22 [█████████████████████-------------------] 52% [02m:22s] Processing chr. 22 [██████████████████████------------------] 52% [02m:23s] Processing chr. 22 [██████████████████████------------------] 52% [02m:24s] Processing chr. 22 [██████████████████████------------------] 54% [02m:25s] Processing chr. 22 [██████████████████████------------------] 54% [02m:26s] Processing chr. 22 [██████████████████████------------------] 54% [02m:27s] Processing chr. 22 [███████████████████████-----------------] 55% [02m:28s] Processing chr. 22 [███████████████████████-----------------] 55% [02m:29s] Processing chr. 22 [███████████████████████-----------------] 55% [02m:30s] Processing chr. 22 [███████████████████████-----------------] 56% [02m:31s] Processing chr. 22 [███████████████████████-----------------] 56% [02m:32s] Processing chr. 22 [███████████████████████-----------------] 57% [02m:33s] Processing chr. 22 [███████████████████████-----------------] 57% [02m:34s] Processing chr. 22 [███████████████████████-----------------] 57% [02m:35s] Processing chr. 22 [████████████████████████----------------] 58% [02m:36s] Processing chr. 22 [████████████████████████----------------] 58% [02m:37s] Processing chr. 22 [████████████████████████----------------] 58% [02m:38s] Processing chr. 22 [████████████████████████----------------] 58% [02m:39s] Processing chr. 22 [████████████████████████----------------] 58% [02m:40s] Processing chr. 22 [█████████████████████████---------------] 60% [02m:41s] Processing chr. 22 [█████████████████████████---------------] 60% [02m:42s] Processing chr. 22 [█████████████████████████---------------] 60% [02m:43s] Processing chr. 22 [█████████████████████████---------------] 61% [02m:44s] Processing chr. 22 [█████████████████████████---------------] 61% [02m:45s] Processing chr. 22 [█████████████████████████---------------] 62% [02m:46s] Processing chr. 22 [█████████████████████████---------------] 62% [02m:47s] Processing chr. 22 [█████████████████████████---------------] 62% [02m:48s] Processing chr. 22 [██████████████████████████--------------] 63% [02m:49s] Processing chr. 22 [██████████████████████████--------------] 63% [02m:50s] Processing chr. 22 [██████████████████████████--------------] 63% [02m:51s] Processing chr. 22 [██████████████████████████--------------] 64% [02m:52s] Processing chr. 22 [██████████████████████████--------------] 64% [02m:53s] Processing chr. 22 [███████████████████████████-------------] 65% [02m:54s] Processing chr. 22 [███████████████████████████-------------] 65% [02m:55s] Processing chr. 22 [███████████████████████████-------------] 65% [02m:56s] Processing chr. 22 [███████████████████████████-------------] 66% [02m:57s] Processing chr. 22 [███████████████████████████-------------] 66% [02m:58s] Processing chr. 22 [███████████████████████████-------------] 67% [02m:59s] Processing chr. 22 [███████████████████████████-------------] 67% [03m:00s] Processing chr. 22 [███████████████████████████-------------] 67% [03m:01s] Processing chr. 22 [████████████████████████████------------] 68% [03m:02s] Processing chr. 22 [████████████████████████████------------] 68% [03m:03s] Processing chr. 22 [████████████████████████████------------] 69% [03m:04s] Processing chr. 22 [████████████████████████████------------] 69% [03m:05s] Processing chr. 22 [████████████████████████████------------] 69% [03m:06s] Processing chr. 22 [████████████████████████████------------] 69% [13m:36s] Processing chr. 22 [█████████████████████████████-----------] 70% [13m:37s] Processing chr. 22 [█████████████████████████████-----------] 70% [13m:38s] Processing chr. 22 [█████████████████████████████-----------] 71% [13m:39s] Processing chr. 22 [█████████████████████████████-----------] 71% [13m:40s] Processing chr. 22 [█████████████████████████████-----------] 71% [13m:41s] Processing chr. 22 [█████████████████████████████-----------] 72% [13m:42s] Processing chr. 22 [█████████████████████████████-----------] 72% [13m:43s] Processing chr. 22 [██████████████████████████████----------] 73% [13m:44s] Processing chr. 22 [██████████████████████████████----------] 73% [13m:45s] Processing chr. 22 [██████████████████████████████----------] 73% [13m:46s] Processing chr. 22 [██████████████████████████████----------] 74% [13m:47s] Processing chr. 22 [██████████████████████████████----------] 74% [13m:48s] Processing chr. 22 [███████████████████████████████---------] 75% [13m:49s] Processing chr. 22 [███████████████████████████████---------] 75% [13m:50s] Processing chr. 22 [███████████████████████████████---------] 75% [13m:51s] Processing chr. 22 [███████████████████████████████---------] 76% [13m:52s] Processing chr. 22 [███████████████████████████████---------] 76% [13m:53s] Processing chr. 22 [███████████████████████████████---------] 77% [13m:54s] Processing chr. 22 [███████████████████████████████---------] 77% [13m:55s] Processing chr. 22 [███████████████████████████████---------] 77% [13m:56s] Processing chr. 22 [████████████████████████████████--------] 78% [13m:57s] Processing chr. 22 [████████████████████████████████--------] 78% [13m:58s] Processing chr. 22 [████████████████████████████████--------] 79% [13m:59s] Processing chr. 22 [████████████████████████████████--------] 79% [14m:00s] Processing chr. 22 [█████████████████████████████████-------] 80% [14m:01s] Processing chr. 22 [█████████████████████████████████-------] 80% [14m:02s] Processing chr. 22 [█████████████████████████████████-------] 80% [14m:03s] Processing chr. 22 [█████████████████████████████████-------] 81% [14m:04s] Processing chr. 22 [█████████████████████████████████-------] 81% [14m:05s] Processing chr. 22 [█████████████████████████████████-------] 82% [14m:06s] Processing chr. 22 [█████████████████████████████████-------] 82% [14m:07s] Processing chr. 22 [█████████████████████████████████-------] 82% [14m:08s] Processing chr. 22 [██████████████████████████████████------] 83% [14m:09s] Processing chr. 22 [██████████████████████████████████------] 83% [14m:10s] Processing chr. 22 [██████████████████████████████████------] 84% [14m:11s] Processing chr. 22 [██████████████████████████████████------] 84% [14m:12s] Processing chr. 22 [██████████████████████████████████------] 84% [14m:13s] Processing chr. 22 [███████████████████████████████████-----] 85% [14m:14s] Processing chr. 22 [███████████████████████████████████-----] 85% [14m:15s] Processing chr. 22 [███████████████████████████████████-----] 86% [14m:16s] Processing chr. 22 [███████████████████████████████████-----] 86% [14m:17s] Processing chr. 22 [███████████████████████████████████-----] 87% [14m:18s] Processing chr. 22 [███████████████████████████████████-----] 87% [14m:19s] Processing chr. 22 [███████████████████████████████████-----] 87% [14m:20s] Processing chr. 22 [████████████████████████████████████----] 88% [14m:21s] Processing chr. 22 [████████████████████████████████████----] 88% [14m:22s] Processing chr. 22 [████████████████████████████████████----] 89% [14m:23s] Processing chr. 22 [████████████████████████████████████----] 89% [15m:50s] Processing chr. 22 [████████████████████████████████████----] 89% [15m:51s] Processing chr. 22 [████████████████████████████████████----] 89% [15m:52s] Processing chr. 22 [█████████████████████████████████████---] 90% [15m:53s] Processing chr. 22 [█████████████████████████████████████---] 90% [15m:54s] Processing chr. 22 [█████████████████████████████████████---] 90% [15m:55s] Processing chr. 22 [█████████████████████████████████████---] 91% [15m:56s] Processing chr. 22 [█████████████████████████████████████---] 91% [15m:57s] Processing chr. 22 [█████████████████████████████████████---] 92% [15m:58s] Processing chr. 22 [█████████████████████████████████████---] 92% [15m:59s] Processing chr. 22 [█████████████████████████████████████---] 92% [16m:00s] Processing chr. 22 [██████████████████████████████████████--] 93% [16m:01s] Processing chr. 22 [██████████████████████████████████████--] 93% [16m:02s] Processing chr. 22 [██████████████████████████████████████--] 94% [16m:03s] Processing chr. 22 [██████████████████████████████████████--] 94% [16m:04s] Processing chr. 22 [██████████████████████████████████████--] 94% [16m:05s] Processing chr. 22 [███████████████████████████████████████-] 95% [16m:06s] Processing chr. 22 [███████████████████████████████████████-] 95% [16m:07s] Processing chr. 22 [███████████████████████████████████████-] 96% [16m:08s] Processing chr. 22 [███████████████████████████████████████-] 96% [16m:09s] Processing chr. 22 [███████████████████████████████████████-] 97% [16m:10s] Processing chr. 22 [███████████████████████████████████████-] 97% [16m:11s] Processing chr. 22 [███████████████████████████████████████-] 97% [16m:12s] Processing chr. 22 [████████████████████████████████████████] 98% [16m:13s] Processing chr. 22 [████████████████████████████████████████] 98% [16m:14s] Processing chr. 22 [████████████████████████████████████████] 99% [16m:15s] Processing chr. 22 [████████████████████████████████████████] 99% [16m:16s] Processing chr. 22 [████████████████████████████████████████] 99% [16m:17s] Processing chr. 22 [████████████████████████████████████████] 100% [16m:19s] Reads simulated
head(seq_results$mutations)
#> chr chr_pos ref alt causes classes S1.occurrences S1.coverage S1.VAF
#> 1 22 16050185 A C B driver 0 26 0.0000000
#> 2 22 16052080 G A germinal 13 33 0.3939394
#> 3 22 16052167 A AAAAC germinal 12 30 0.4000000
#> 4 22 16052986 C A germinal 20 40 0.5000000
#> 5 22 16053444 A T germinal 18 30 0.6000000
#> 6 22 16053659 A C germinal 35 35 1.0000000
#> S2.occurrences S2.coverage S2.VAF S3.occurrences S3.coverage S3.VAF
#> 1 0 30 0.0000000 26 42 0.6190476
#> 2 25 37 0.6756757 24 38 0.6315789
#> 3 27 43 0.6279070 11 28 0.3928571
#> 4 11 27 0.4074074 16 33 0.4848485
#> 5 16 26 0.6153846 19 33 0.5757576
#> 6 32 32 1.0000000 24 24 1.0000000ProCESS allows to visualise the sequencing results.
f_seq <- seq_results$mutations %>% filter(classes != "germinal")
plot_VAF_histogram(f_seq, labels = f_seq["classes"], cuts = c(0.02, 1))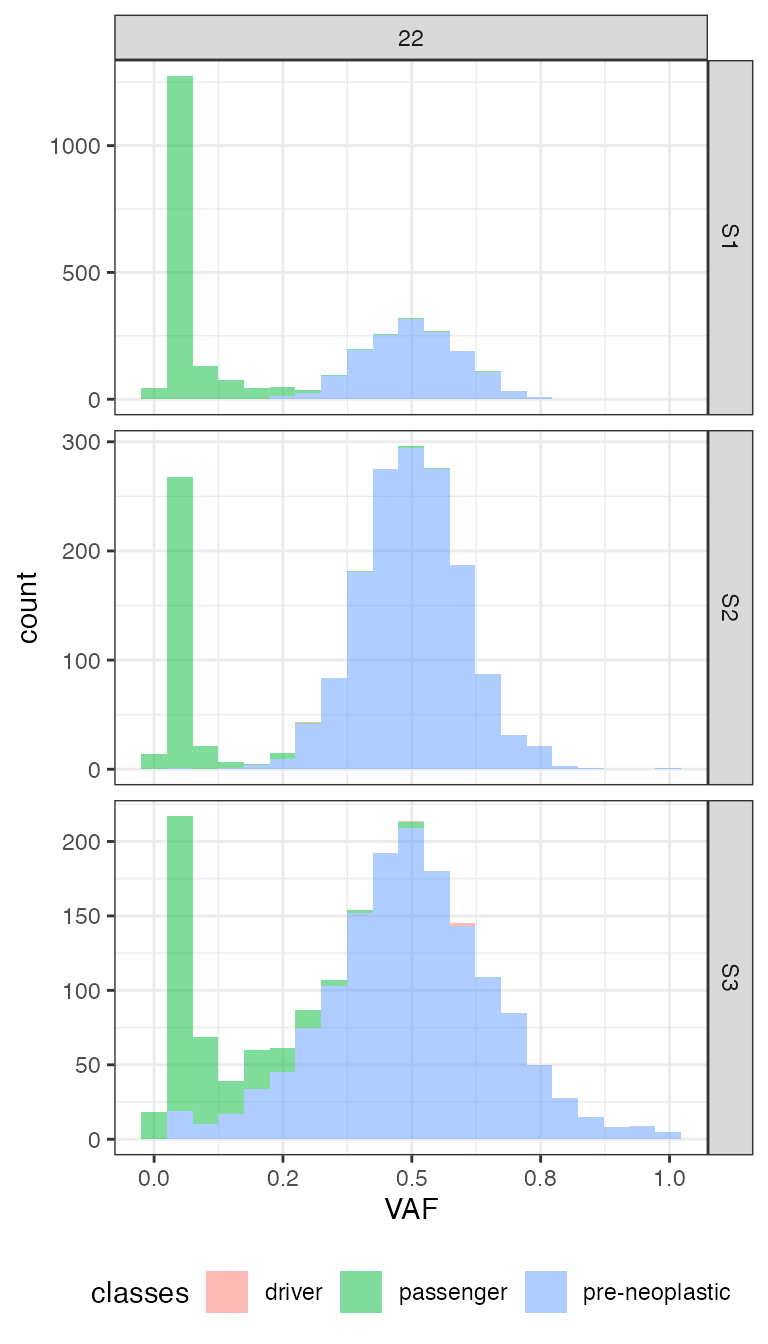
Marginal distributions can also be plotted.
plot_VAF_marginals(f_seq, samples = c("S1", "S2", "S3"),
labels = f_seq["classes"])
#> [[1]]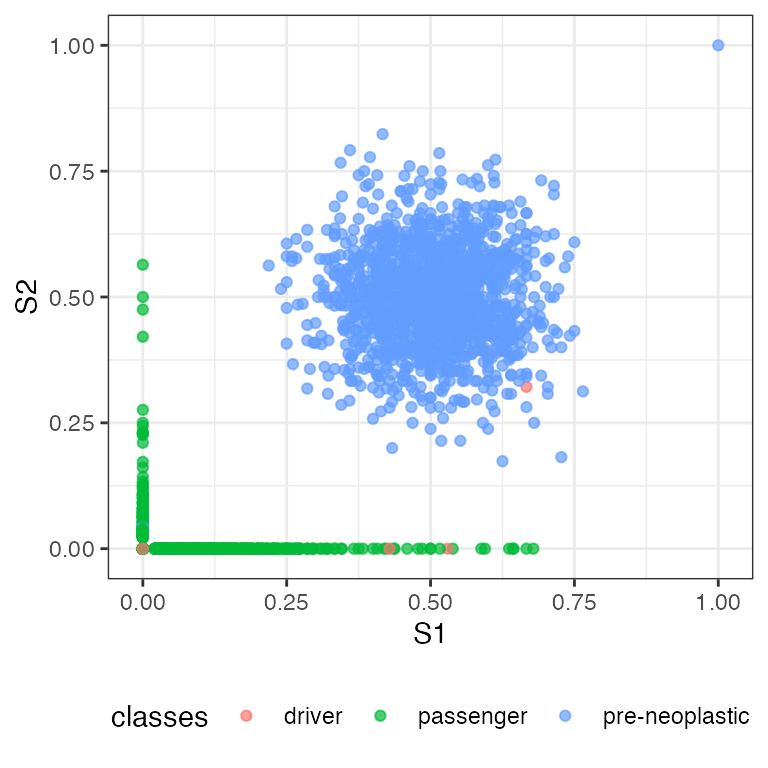
#>
#> [[2]]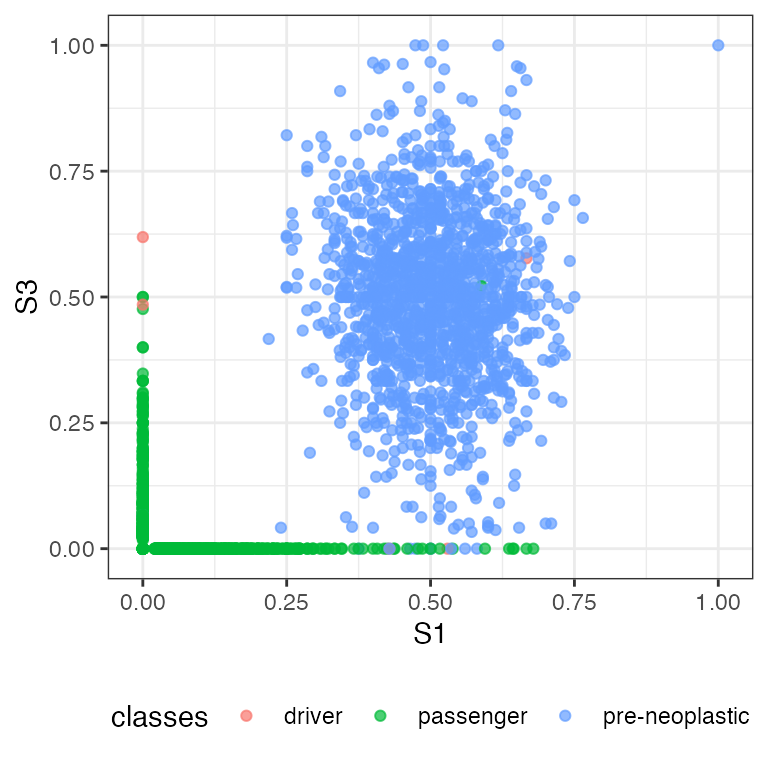
#>
#> [[3]]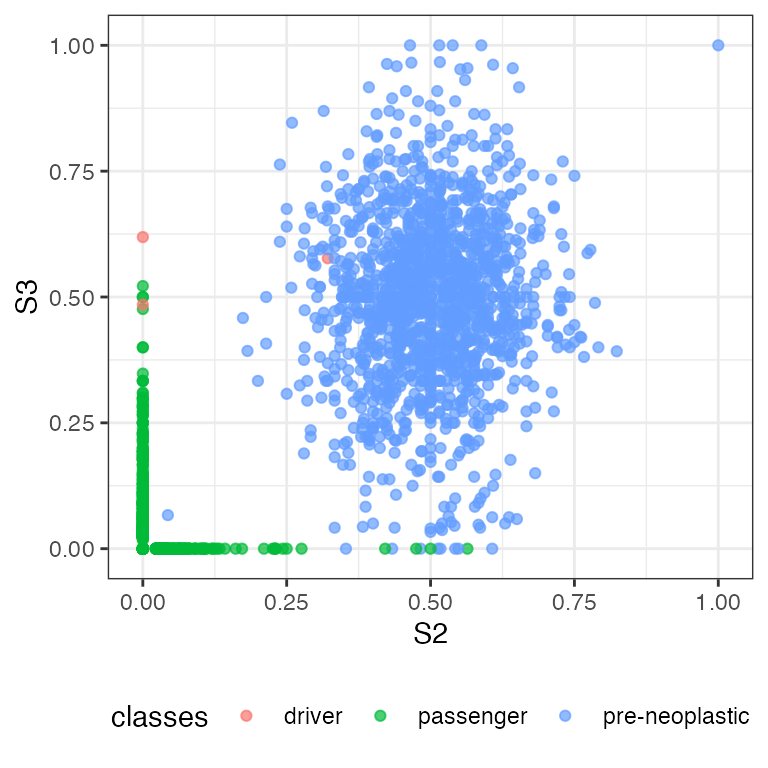
In S1 vs S3 figure, we can some spot passenger mutations occurring in both S1 and S3. Let us identify these mutations.